Reprocesamiento de los datos
A partir de la pagina web de Git proMGE realizamos modificaciones de los scripts para mejorar la documentación de los mismos.
Datos
Modificado por: Evelia Coss
Todos los datos utilizados en este estudio están publicados y a disposición del público, como se indica en la sección de material y métodos del manuscrito.
- Los datos de todos los genomas procariotas utilizados en el estudio están disponibles en https://progenomes.embl.de/.
- La información sobre el hábitat está disponible en GMGC.
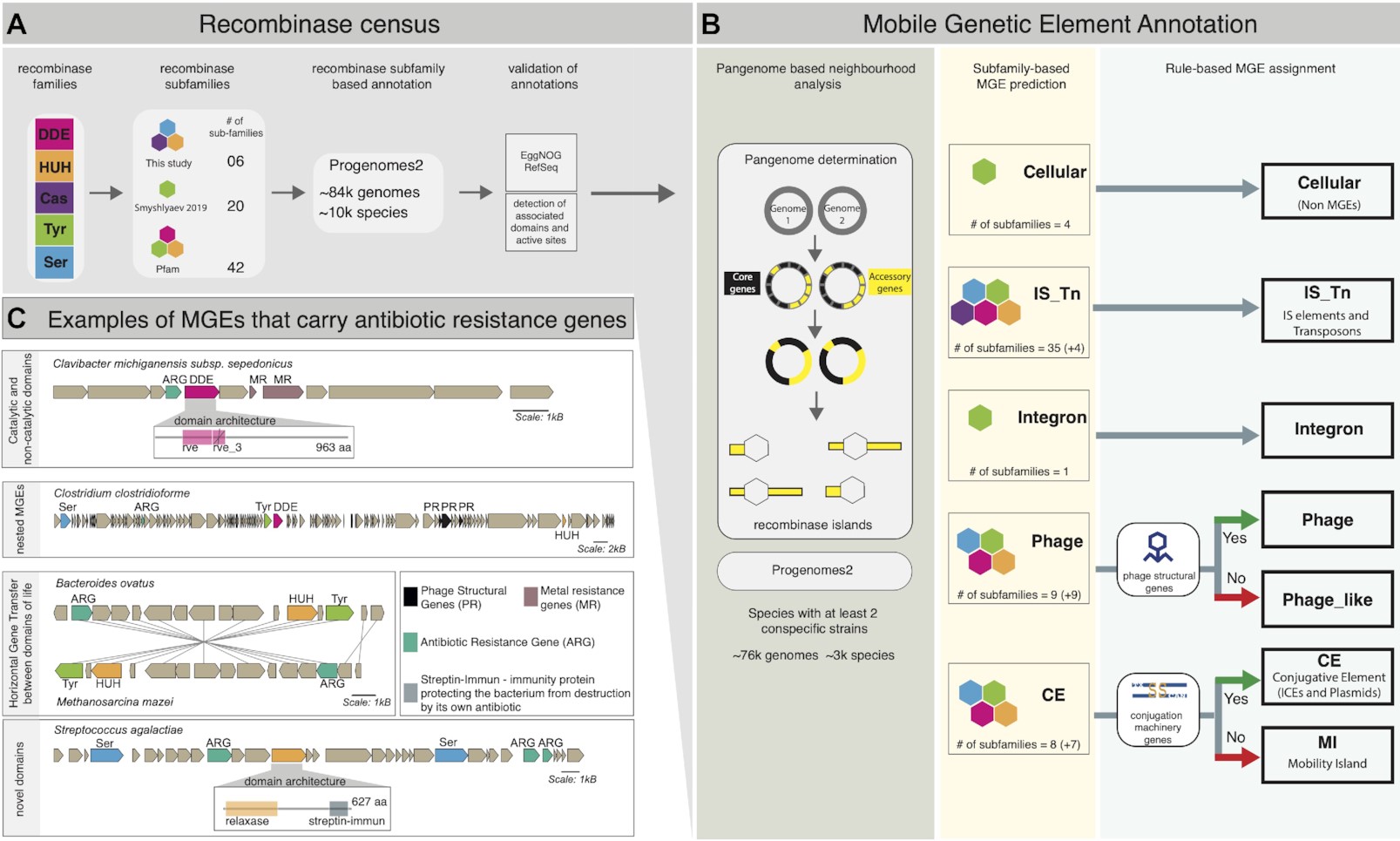
Procesamiento de datos para la Figuras
Los autores nos informan que se realizaron modificacione sobre los inputs para generar las Figuras 2 a 5 empleando codigo de perl. Para mas informacion vista la fuente original del Git.